Gips kommt in erheblichen Mengen in der Natur in Lagerstätten als Calciumsulfat Dihydrat vor. Durch Erhitzen wird u.a. daraus das Calciumsulfat Hemihydrat hergestellt. Dieses wird vielfältig als u.a. als Baustoff, zum Abformen und Modellieren sowie in Gipsverbänden eingesetzt. Dazu wird das Calciumsulfat Hemihydrat mit Wasser versetzt. Im Umgangs mit dem Pulver sollte Staubentwicklung vermieden werden. Zum kreativen Gestalten und Experimentieren ist Hobby- und Modelliergips u.a. in Baumärkten erhältlich, ein Sicherheitsdatenblatt zum Produkt z.B. hier ….
Aus zwei Volumenanteilen Gipspulver und einen Volumenanteil Wasser wird ein dünnflüssige Suspension hergestellt. In der wässrigen Suspension entsteht aus dem Hemihydrat das Dihydrat, dabei entsteht nach einiger Zeit unter Abgabe von Wärme fester Gips.
Zur Temperaturmessung wurde der Sensor Go!Temp von Vernier eingesetzt.
Bezugsquelle für den Sensor in Deutschland: LPE Naturwissenschaft & Technik GmbH
Der Sensor kann am USB-Anschluss eines Rechners bzw. am LabQuest 2 eingesetzt werden.Der Sensor ist relativ robust, daher kann er auch als Rührstab eingesetzt ( dazu sollten die klassischen Thermometer aus Glas nicht verwendet werden ).
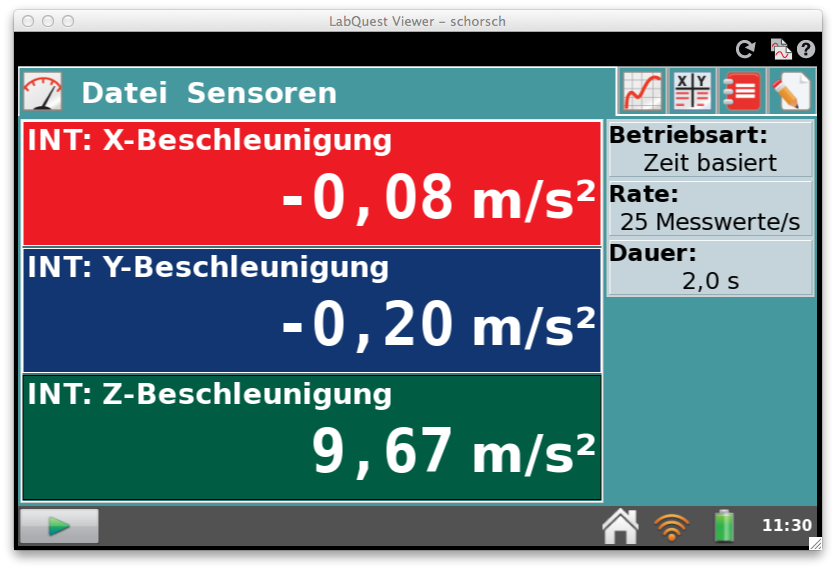
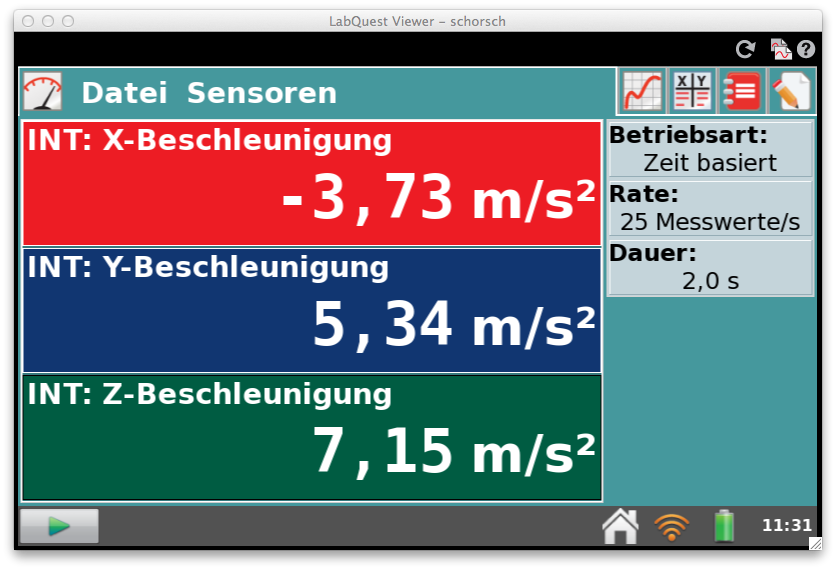
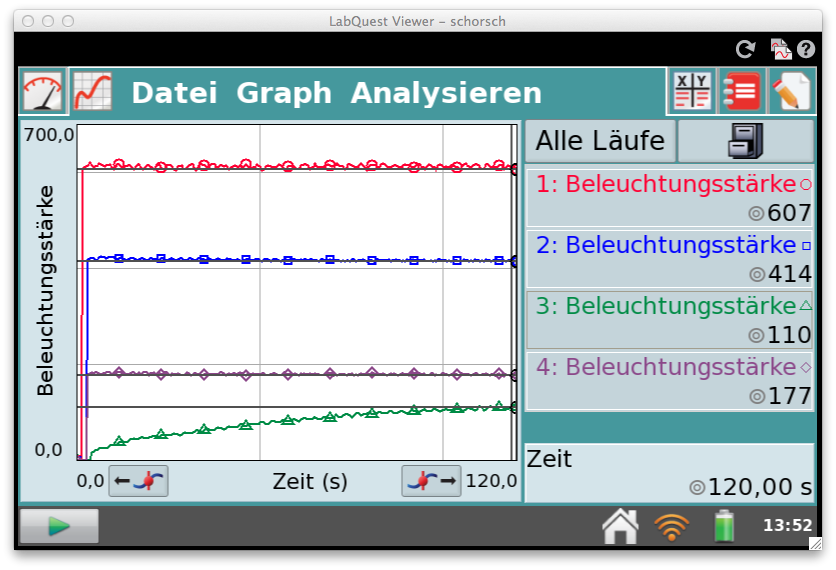
Hier wurde der Sensor am LabQuest 2 angeschlossen. Über WLAN wurde eine Verbindung zum Mac Book hergestellt und dort mit dem LabQuest Viewer und dem Fototool der Smart Notebook Software die folgenden Bildschirmfotos aufgenommen.
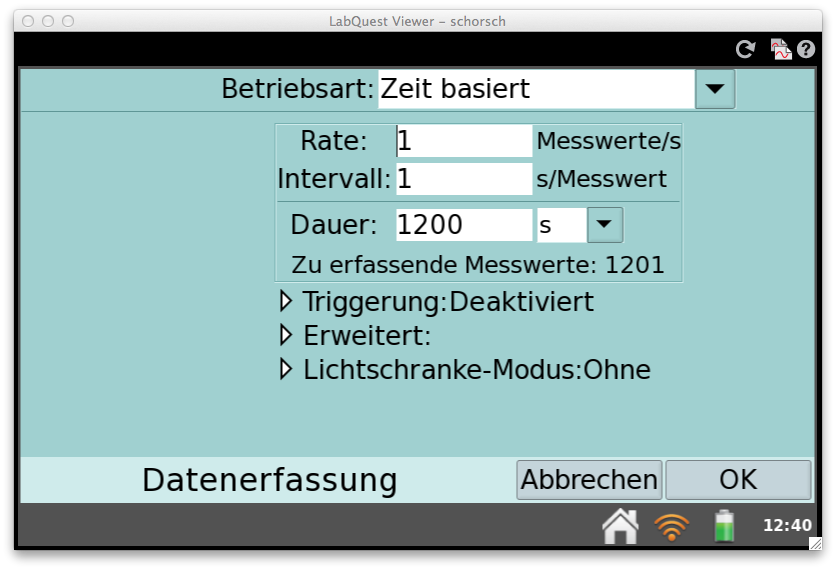
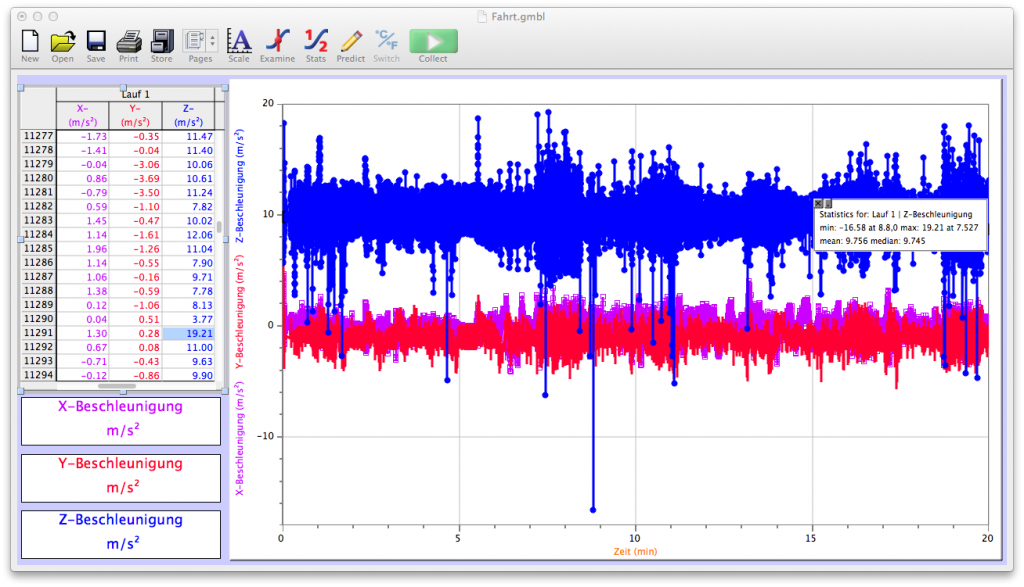
Die ursprünglich gewählte Messzeit war zu kurz.
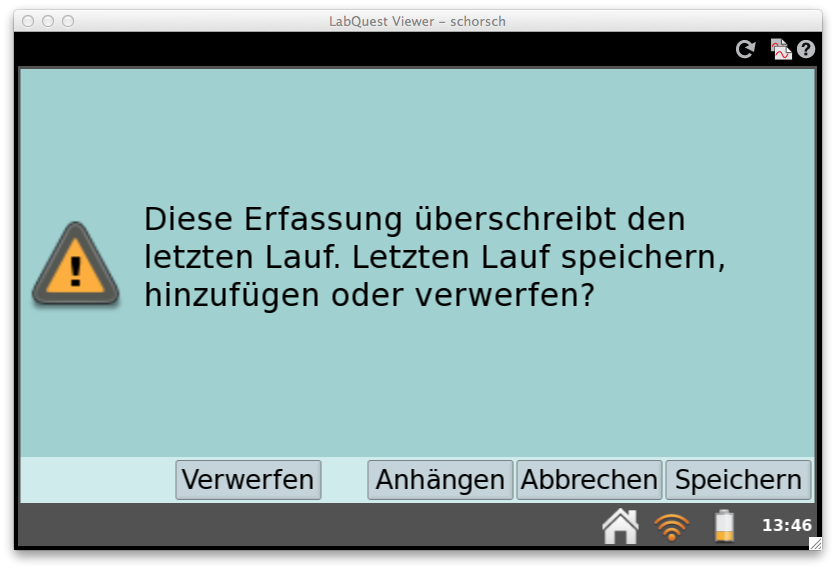
Daher wurde die Messung mehrfach fortgesetzt und an die Messreihe angehängt.
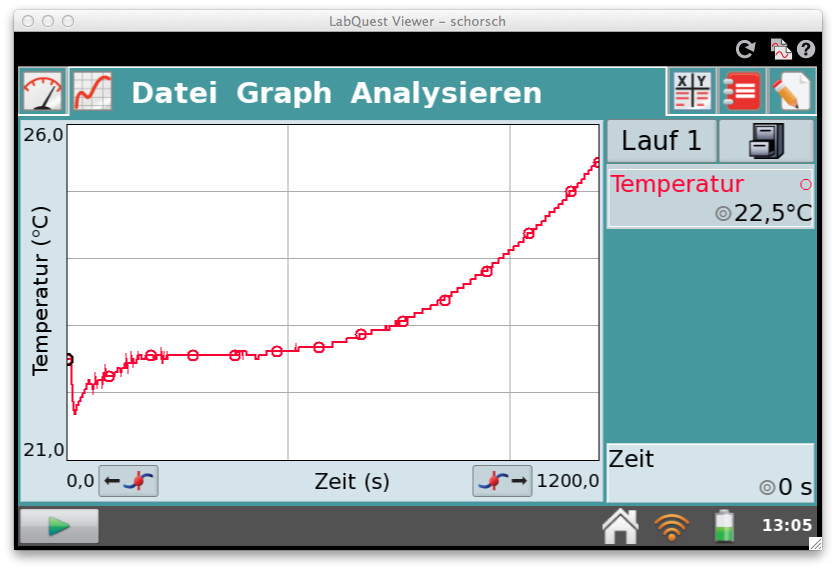
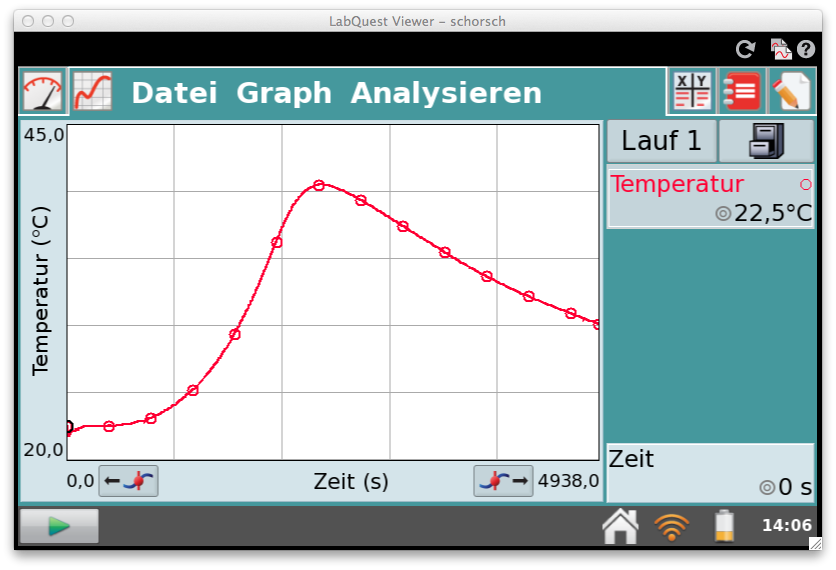
Nach viermaligem Durchführen der Messung – die Zeit der Messpausen wurde in die Datenreihe einbezogen – daher sind keine „Sprünge“ im Kurvenverlauf sichtbar.
Am Kurvenverlauf sowie in der Sinnlichen Wahrnehmung kann beim Versuch der Reaktionsverlauf, das Reaktionsende und das Abkühlen beoachtet werden.
Der Sensor kann problemlos aus dem festen jedoch noch feuchten Gips gezogen und mit einem feuchten Tuch gereinigt werden.