Früher benötigte man zur Darstellung von Molekülstrukturen eine „Grafik-Workstation“ und die Einarbeitung in komplexe Programme, zu deren Bedienung über ein Terminal Skripte eingegeben werden mussten.
Es ist daher sehr beachtlich was nun mit geeigneten Apps und dem iPAD intuitiv möglich ist.
Es wird hier kurz auf zwei kostenfreie und eine kostenpflichtige App eingegangen.
Die Bildschirmfotos können durch anklicken vergrößert werden.
Die Daten des dargestellten Proteins finden Sie hier:
http://www.rcsb.org/pdb/explore/explore.do?structureId=1QI9
Nun zu den Apps
„Molecules“ von Sunset Lake Software
Zitat aus der Bewertung zur App:
“ Die ganze Idee fantastisch, aber die Deutsche Sprache fehlt. Bitte ändern!“
Dieser Wunsch ist sinnfrei. Die App biete den Zugang zu internationalen Strukturdatenbanken, Grundkenntnise der englischen Fachsprache sind nötig um in diese Datenbeständen sinnvoll zu nutzen.
Zitat aus der Beschreibung der App:
„…structures can be viewed in both ball-and-stick and spacefilling visualization modes ..“
Damit eignet sich die App vorrangig zur Darstellung kleiner Moleküle, den Zugang zu diesen Strukturen erhält man direkt aus dem Suchfeld der App über: http://pubchem.ncbi.nlm.nih.gov/
Zur Suche benötigt man den englischen Namen der Substanz oder deren Summenformel.
Aus der App kann man auch nach Proteinstrukturen suchen. Die Daten stehen hier zur Verfügung: http://www.rcsb.org/pdb
Die Darstellung der Proteine liefert eine Übersicht der räumlichen Form, jedoch keine weiteren Informationen und Einblicke in die Strukturen.
Für diesen Zweck ist die App „RCSB PDB Mobile“ von RCSB PDB wesentlich besser geeignet.
„The PDB was established in 1971 at Brookhaven National Laboratory and originally contained 7 structures.“
Mehr zur Geschichte der Datenbank hier: http://journals.iucr.org
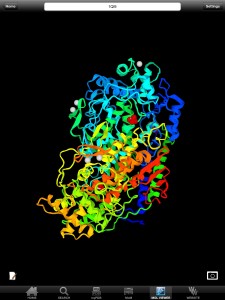
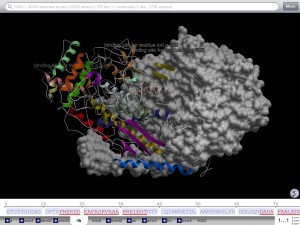
Darstellung: „ribbon“ zeigt Sekundär- und Tertär-Struktur
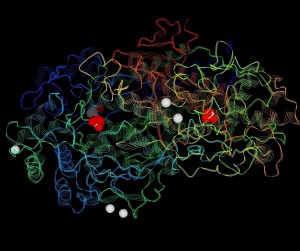
Darstellung: Proteinstruktur als „strands“ – Nichtproteinteile sind als „spacefilling“ dargstellt, damit sind in den beiden Untereinheiten jeweils ein Vanadation ( mit – 4x rot und 1x weiß ) im aktiven Zentrum sichtbar. An der Oberfläche ( weiß ) sind Iodatome zu finden.
Die App ist nicht nur ein Molekül-Viewer, sondern bietet vielfältige Zugangsmöglichkeiten zu Informationen über Proteine und anderen makromolekularen Strukturen.
Zum Stöbern in den Strukturen läd das „Molecule of the Month Archive“ ein.
Ein Vorschlag zum Start: PDB Pioneers
Zu diesen Pionieren gehören die Nobelpreisträger für Chemie des Jahres 1962.
The Nobel Prize in Chemistry 1962 was awarded jointly to Max Ferdinand Perutz and John Cowdery Kendrew „for their studies of the structures of globular proteins“
Neben diesen beiden kostenlosen Apps gibt es verschiedene andere hier soll kurz auf eine kostenpflichtige App eingegangen werden: „iMolview“ von Molsoft
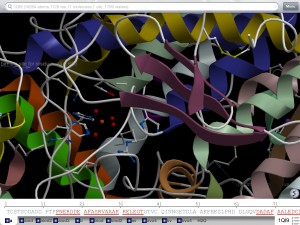
Darstellung: Untereinheit a – „ribbons“ – Untereinheit b – Oberfläche der Struktur – es wird am unteren Rand die Sequenz angezeigt.
Blick ins aktive Zentrum der Untereinheit a – u.a. zwischen den Sekundärstrukturelementen zwei Histidine und das Vanadation ( rote Punkte ) sichtbar.
Zitat aus der Beschreibung der App:
„iMolview is an app for the iPhone/iPad and Android that lets you browse protein, DNA, and drug molecules in 3D. The app has a direct link to the Protein Data Bank (PDB) and DrugBank and has a fast and easy to use interface. Touching the molecules via the screen allows you to interact immediately with the 3D structures in a unique way. You can zoom in and out, rotate, spin, pan, and clip the 3D molecules with your finger tips in ways that are impossible using a traditional mouse and desktop computer.“
Mit allen genannten Apps können Strukturen auch aus anderen Quellen beim Surfen mit dem Browser über den Dialog „öffnen in“ dargestellt werden
Die gespeicherten Strukturdaten können u.a. über itunes zwischen iPAD und Mac oder PC ausgetauscht werden.